| Lingua : |
|
| Comunità Encyclopedia |Enciclopedia Risposte |Invia domanda |Conoscenza Vocabolario |Carica conoscenza |
Ibridazione in situ fluorescente |
 |
|
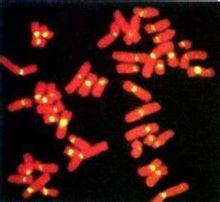
Ibridazione in situ fluorescente Breve introduzione Fluorescence in situ hybridization metodo è una mappa fisica metodo di disegno, usando la sonda marcato con fluoresceina per rilevare la sonda e cromosomi in metafase o interfase cromatina ibridazione. Ibridazione in situ fluorescente (fluorescenza in situ ibridazione, FISH) alla fine del 1980, radioattivo nelle tecniche di ibridazione in situ sviluppate sulla base di un non-radioattivo tecniche di citogenetica molecolare per sostituire il marcatore fluorescente formata una marcati con isotopi La nuova specie di ibridazione in situ, la sonda associata a Il principio di base prima molecola-mediata (giornalista molecola) con il processo ibrido poi attraverso il immunocitochimica colorante fluorescente connessione. pesce è il DNA (o RNA) con sonde specifici marcatori molecolari nucleotidiche, e l'ibridazione della sonda direttamente nel cromosoma o DNA fette fibra e poi con fluoresceina monoclonali coniugati sonde molecola di anticorpo per rilevare molecola specifica sequenza di legame del DNA cromosomico o fibre di DNA fetta qualitativo, posizionamento, analisi quantitativa relativa. FISH è sicuro, veloce, ad alta sensibilità, la sonda può essere conservato a lungo in grado di visualizzare una varietà di colori, ecc, non solo in grado di visualizzare metafase, ma anche dimostrato nel nucleo interfase. mentre sulla base di ibridazione in situ fluorescente e lo sviluppo di una fluorescenza multicolore ibridazione in situ e cromatina fibra ibridazione in situ fluorescente.Sfondo Application RRNA per l'uso di ibridazione in situ fluorescente, le seguenti cause possono portare ad una intensità di segnale di fluorescenza inferiore: Contenuto ribosomi inferiori Minore permeabilità delle cellule circostanti La sequenza bersaglio può essere un contatto inferiore (a causa della conformazione piegatura rRNA, alcuni degli altri posizione e la molecola di rRNA o catena proteica o altro rRNA stretto contatto, in modo che la sonda e la sequenza bersaglio ibridazione può) Al fine di verificare se la sequenza della cellula bersaglio è facile ibridazione, e il test ottimale temperatura di ibridazione può essere usato per "clonare ibridazione in situ fluorescente" (clone-FISH) prova: il gene rRNA incorporato nel plasmide è stato trasformato in Escherichia coli espressione che costituisce il ribosoma, e sonde poi fluorescente. FISH può essere combinato con la citometria a flusso, microscopio a fluorescenza con conta delle cellule specifiche o di separazione. [1] Varianti Fluorescenza Enzyme-linked ibridazione in situ (CARD-FISH) Tecniche di ibridazione in situ fluorescente (FISH) dettagliate 1974 sarà la prima volta Evans cromosoma tecniche di bendaggio e l'ibridazione in situ in combinazione, migliorare la precisione di posizionamento. Alla fine del 1970, la gente ha cominciato ad esplorare la ibridazione fluorescente in situ, cioè tecnologie FISH. 1981 Harper è riuscito a sequenze di DNA a singola copia mirati agli esemplari G-banding, segnando la tecnologia di localizzazione cromosomica ha compiuto importanti progressi. Nel 1990, con la conduzione del progetto genoma umano, perché la mappatura ad alta risoluzione delle esigenze del genoma umano, la tecnologia FISH è stato rapido sviluppo e l'applicazione larga. Principio FISH tecnica (ibridazione in situ fluorescente) è un importante non radioattivo ibridazione in situ. Il suo principio di base è: se il cromosoma fette rilevato o fibre di DNA con l'utilizzo del DNA bersaglio è omologa a una sonda di acido nucleico complementare, i due denaturato - Ricottura - rinaturazione può essere formata con la sonda di acido nucleico DNA bersaglio un ibrido. Il rapporto sonda di acido nucleico su un particolare molecole marcate nucleotidiche come biotina, digossina, può utilizzare il rapporto marcato con fluoresceina molecole con avidina tra specifica reazione immunochimica, il sistema di rilevazione di fluorescenza allo specchio DNA da misurare sotto il, analisi quantitativa o qualitativa posizionamento relativo. Procedura sperimentale Preparazione del campione FISH → → Preparazione sonde di ibridazione sonda marcata → → (cromosoma banding) → → risultati delle analisi microscopio a fluorescenza. Caratteristica In situ sonde di ibridazione divisi per tipo di molecole marcatore radiomarcati e l'etichettatura non radioattivo. Marcato con un isotopo radioattivo vantaggio sonda di meno pesante per la preparazione dei campioni, è possibile estendere il tempo di esposizione per migliorare la potenza del segnale, è più sensibile. Svantaggio è che la sonda è instabile, autoradiografia lungo tempo, la diffusione delle radiazioni rende risoluzione spaziale non è elevato, e isotopi funzionamento più complicato e così via. Sistema di marcatura fluorescente può essere utilizzato per risolvere tali inconvenienti, questa è la tecnica FISH. Tecnica FISH come un sistema di rilevamento non radioattivo ha le seguenti caratteristiche. Vantaggi: 1, reagenti fluorescenti e sonde economiche, di sicurezza; 2, la stabilità della sonda, un marchio entro due anni dopo l'uso; 3, periodo di sperimentazione breve, possono ottenere rapidamente risultati, specificità, precisione di posizionamento, 4, pesce può trova nella 1kb lunghezza sequenza di DNA, la sensibilità di sonde radioattive considerevoli; 5, multicolor FISH nello stesso nucleo attraverso colori diversi possono essere visualizzati simultaneamente rilevare sequenza multipla; 6, sia nella presentazione, o il numero di cromosomi in metafase cambiamenti nella struttura possono essere visualizzati anche nell'interfase struttura del DNA cromosomico sospensione. Svantaggi: non raggiungere il 100% ibridazione, soprattutto nell'applicazione di una breve sonde di cDNA diminuito significativamente l'efficienza. Applicazione La tecnologia può essere utilizzata non solo in una sequenza di geni noti o localizzazione cromosomica, ma anche può essere usato per clonare i geni o marcatori genetici non ha e lo studio delle aberrazioni cromosomiche. In gene qualitativi, quantitativi, l'integrazione, e di altri aspetti dello studio hanno espresso notevole vantaggio. FISH originariamente utilizzato per metafase i cromosomi. Differenziate da essere preparati in questo nucleo è cromosomi altamente condensati, ogni cromosoma ha una forma riconoscibile, mostreranno una colorazione caratteristica della posizione del centromero e colorazione di tipo band. Quando si tratta di cromosomi in metafase, FISH ottenuto misurando il segnale di fluorescenza rispetto alla posizione dell'estremità del braccio corto del cromosoma (valore FLpter) da tracciare. Cromosomi in metafase con carenze che, dal momento che la natura del suo, la mappatura solo a bassa risoluzione altamente condensato, almeno due indicatori separati a 1MB segnale di ibridazione come un separato distinguibile (Trask et al., 1991) . Questa risoluzione non è abbastanza per costruire un cromosoma scambi mappe. Pertanto metafase cromosoma FISH è usato principalmente per identificare nuovi marcatori su cromosomi posizione approssimativa, per altri metodi di mappatura più sofisticati da preparare. Tutti insieme, questi "altri metodi" non include alcuna di pesce, ma nel 1995, dopo una serie di tecnica FISH ad alta risoluzione è stato sviluppato. Queste tecnologie essere studiate variando la natura del preparato di cromosomi per ottenere una risoluzione più alta. Metafase i cromosomi si condensano per la mappatura fine è troppo alto, quindi abbiamo bisogno di utilizzare più cromosomi stirata. Ci sono due modi per soddisfare questo requisito (Heiskanen et al, 1996.): Cromosoma tratto meccanica (cromosoma allungato meccanicamente) cambiando il supporto per condividere il nucleo ottenuto mediante il metodo cromosomi. Shearing forza centrifuga può essere allungato alla lunghezza normale dei cromosomi di 20. Ogni cromosoma può ancora essere identificato sui metodi di mappatura segnale di pesce tipicamente affrontare gli stessi cromosomi in metafase, in modo che la risoluzione può essere notevolmente migliorata, in grado di distinguere oltre 200 ~ segno di 300kb. Cromosomi non metafase (cromosoma non-metafase) solo a metà altezza dei cromosomi condensati, mentre le altre fasi del ciclo cellulare per mantenere lo stato naturale di non confezionati, i ricercatori hanno usato-nucleo pre, tempo di condensazione cromosomica sufficiente per distinguere i singoli cromosomi. In pratica, questo metodo non è superiore ad meccaniche posto di stretching cromosomi. In contrasto interfase (interfase) cromosomi sono più utili perché Interfase (divisione nucleare di nuovo tra i) il livello più basso degli imballaggi cromosoma. Utilizzare cromosomi interfase, la risoluzione può raggiungere 25kb o meno, ma cromosoma morfologia scomparso, posizionando la posizione della sonda per promuovere la necessità di punto di riferimento esterno. Pertanto, la tecnologia può essere approssimativamente mappatura cromosomica è stata usata dopo, di solito come una piccola area per determinare ordine cromosomico di una serie di marcatori. Cromosomi interfase contengono tutte le celle all'assemblaggio di molecole di DNA. Per migliorare ulteriormente la risoluzione di FISH a 25kb o meno, è necessario dare un cromosoma completo, l'uso di DNA purificato. Questo metodo viene chiamato in fibra FISH (fibra-FISH), utilizzando la tecnologia gel pettinatura molecolare per preparare allungato o DNA, in grado di distinguere la distanza di voto inferiore a 10kb. [2] Fluorescenza in situ processo di sviluppo della tecnologia di ibridazione FISH per rilevare il numero di siti e lo sviluppo di rilevamento del bersaglio Dopo la tecnica FISH fondo istituito, FISH è non solo per singolo gene o rilevamento dell'acido nucleico, tecnologia FISH estende per l'ulteriore sviluppo di multicolore FISH rilevamento simultaneo di più loci genici, dallo sviluppo di test genetici nel genoma, cromosomi, nelle cellule viventi mRNA trascrizione originale rilevazione bit e acido nucleico livello di rilevamento di organizzazione, e in studi futuri possono essere applicate anche al rilevamento di tutto l'organismo. Primi sonde maggiore proliferazione dal vettore, metodo nick translation, nel metodo di trascrizione in vitro e il metodo random primer per preparare sintesi del DNA per ottenere la clonazione ibridazione specifica. Tuttavia, di solito con un grande frammento delle ripetizioni sonda causando sfondo un'elevata fluorescenza, utilizzando senza etichetta pretrattamento acido nucleico a siti di inibizione vincolanti e non specifico di ibridazione non specifica può essere utilizzato per superare i problemi di cui sopra, ma permette anche ai ricercatori di ampliare rilevare il bersaglio, per realizzare un intero colorazione cromosomica. Tecnica FISH in analisi citogenetica nel cromosoma quindi stata notevolmente migliorata. Ad esempio, per ibridazione genomica comparativa (ibridazione Comparativegenomic) è utilizzato per rilevare delezioni e duplicazioni di regioni cromosomiche. Una volta che il grande frammento sonda ed il campione formeranno un segnale di legame non specifico, il rilevamento di geni sul cromosoma confusione, dobbiamo tagliare in piccoli frammenti <200 nucleotidi. Metodi di rilevamento migliorati Ora e software di rilevamento che rende la tecnica FISH continua a sviluppare sempre più esigente bassa sensibilità di rilevamento è in aumento. Algoritmi di elaborazione di immagini accurate informatici continuano a migliorare la formazione di livello sonde tecniche ad alta risoluzione submicroscopiche. Con l'obiettivo di rilevazione sempre più piccola, la tecnica FISH è stata utilizzata sotto copertura nucleare riarrangiamento genico subtelomeric e mappatura cromosomica e la rilevazione accurata di singola copia mRNA. FISH espansione della gamma di rilevazione, rendendo l'applicazione della tecnica FISH in rapida crescita degli anni 1990. È formata dalla applicazione della tecnologia FISH per realizzare un crescente rami di molti diversi tipi di loci rilevate simultaneamente. Il primo è l'uso di un numero di rivelazione di fluorescenza differente sempre punti, come la fluorescenza a due colori per il rilevamento di sequenze specifiche di acidi nucleici, ogni cromosoma, gene o prodotti di trascrizione sono state poi fatte di un segnale fluorescente può essere risolto per rappresentare. Dopo la decodifica schema è l'uso di due colori FISH espandere ulteriormente la gamma di applicazioni. Programma di decodifica è principalmente per la percentuale di colore, cioè, ogni colore nella proporzione di colore totale per descrivere più siti. Ciascuno dei metodi di cui sopra, o una combinazione di entrambi gli approcci devono essere rilevati fino a 12 loci. Colorato utilizzando un programma di traduzione computer possa rilevare tutti i cromosomi umani rappresentano loci multipli rilevati da FISH pietra miliare. Anche se è possibile utilizzare una varietà di metodi di mRNA osservati, ma tutta l'analisi FISH trascrizione sembra essere più applicazioni in-situ. Colore tecnica di codifica è stato implementato il rilevamento dell'intera organizzazione. |
| Utente Recensione |
|
Ancora nessun commento |